第一节 WinBUGS数据分析案例
在这一节中,我将拿一个经典的研究数据,利用WinBUGS给出简单的分析。首先介绍一下这个数据:Seeds
seed O. aegyptiaco 75 seed O. aegyptiaco 73 Bean Cucumber Bean Cucumber
r n r/n r n r/n r n r/n r n r/n
10 39 0.26 5 6 0.83 8 16 0.5 3 12 0.25
23 62 0.37 53 74 0.72 10 30 0.33 22 41 0.54
23 81 0.28 55 72 0.76 8 28 0.29 15 30 0.5
26 51 0.51 32 51 0.63 23 45 0.51 32 51 0.63
17 39 0.44 46 79 0.58 0 4 0 3 7 0.43
10 13 0.77
这个数据来自Crowder (1978)。之后Breslow and Clayton (1993) 作为例子,也分析过这个数据。数据反映的是某一品种的豆类种子和某一品种的黄瓜种子,分别放在21个培养皿(plates)中分别培养,在根提取物aegyptiaco 75和aegyptiaco 73的作用下的出芽率的差异。其中r是出芽的个数,n是种子的个数,而r/n是出芽率。我们用random effect logistic regression模型来进行分析(注意,在Bayesian分析中,通常是将covariates看做是服从某一个分布的随机变量,这和一般意义上的GLM, GLME, LME中对于covariates解释是不同的):
\(r_i \sim Binomial(p_i, ~n_i)\)
\(logit(p_i)=\alpha_0 + \alpha_1 x_{1i} + \alpha_2 x_{2i} + \alpha_{12} x_{1i} x_{2i} + b_i\)
\(b_i \sim Normal(0, \tau)\)
其中\(x_{1i}\)是种子的类型,\(x_{2i}\)是根提取物的类型,\(\alpha_{12} x_{1i} x_{2i}\)是交互项, \(\alpha_0,~\alpha_1,~\alpha_2,~\alpha_{12},~\tau\)是给定的独立的 “noninformative” 先验参数。在Bayesian分析中,通常我们会定义一个DAG图(即Directed Acyclic Graph有向无圈图) 。我们可以在WinBUGS中通过设计DAG图来定义模型。不过这一节中我们还是用WinBUGS中的BUGS语言来定义模型,如何在WinBUGS中通过设计DAG图来定义模型我将在下一节中详细介绍,但是必须要说明的是BUGS语言比DAG图灵活,不过直观性不如后者。
模型
model
{
for( i in 1 : N ) {
r[i] ~ dbin(p[i],n[i])
b[i] ~ dnorm(0.0,tau)
logit(p[i]) <- alpha0 + alpha1 * x1[i] + alpha2 * x2[i] +
alpha12 * x1[i] * x2[i] + b[i]
}
alpha0 ~ dnorm(0.0,1.0E-6)
alpha1 ~ dnorm(0.0,1.0E-6)
alpha2 ~ dnorm(0.0,1.0E-6)
alpha12 ~ dnorm(0.0,1.0E-6)
tau ~ dgamma(0.001,0.001)
sigma <- 1 / sqrt(tau)
}
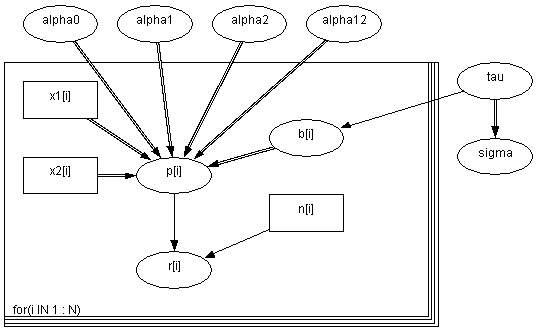 WinBUGS doodle模型
WinBUGS doodle模型
数据
list(r = c(10, 23, 23, 26, 17, 5, 53, 55, 32, 46, 10, 8, 10, 8, 23, 0, 3, 22, 15, 32, 3),
n = c(39, 62, 81, 51, 39, 6, 74, 72, 51, 79, 13, 16, 30, 28, 45, 4, 12, 41, 30, 51, 7),
x1 = c(0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 1, 1, 1, 1, 1, 1, 1, 1, 1, 1),
x2 = c(0, 0, 0, 0, 0, 1, 1, 1, 1, 1, 1, 0, 0, 0, 0, 0, 1, 1, 1, 1, 1),
N = 21)
初始值
list(alpha0 = 0, alpha1 = 0, alpha2 = 0, alpha12 = 0, tau = 1)
经过10000次迭代,我们得到参数的估计如下:
node mean sd MC error 2.50% median 97.50% start
alpha0 -0.5546 0.1941 0.007696 -0.9353 -0.5577 -0.1597 1001
alpha1 0.08497 0.3127 0.01283 -0.5814 0.09742 0.6679 1001
alpha12 -0.8229 0.4321 0.01785 -1.697 -0.8218 0.01641 1001
alpha2 1.356 0.2743 0.01236 0.8257 1.347 1.909 1001
sigma 0.2731 0.1437 0.007956 0.04133 0.2654 0.5862 1001
第二节 结合SAS做比较分析
下面我们用同样的数据在SAS中进行分析,看看结果相差多少:
首先生成数据:
data seeds;
input plate r n seed extract;
datalines;
1 10 39 0 0
2 23 62 0 0
3 23 81 0 0
4 26 51 0 0
5 17 39 0 0
6 5 6 0 1
7 53 74 0 1
8 55 72 0 1
9 32 51 0 1
10 46 79 0 1
11 10 13 0 1
12 8 16 1 0
13 10 30 1 0
14 8 28 1 0
15 23 45 1 0
16 0 4 1 0
17 3 12 1 1
18 22 41 1 1
19 15 30 1 1
20 32 51 1 1
21 3 7 1 1
;
run;
data seeds;
set seeds;
interact=seed*extract;
run;
proc print; run;
然后调用GENMOD过程,拟合经典的logistic regression回归模型
proc genmod data=seeds;
/* Basic logistic regression WHITHOUT random plate effect */
title ‘Classical logistic regression’;
model r/n= seed extract interact / dist=B;
run;
得到
Analysis Of Parameter Estimates
Parameter DF Estimate Standard Error Wald 95% Confidence Limits Chi-Square Pr > ChiSq
Intercept 1 -0.5582 0.126 -0.8052 -0.3112 19.62 <.0001
seed 1 0.1459 0.2232 -0.2915 0.5833 0.43 0.5132
extract 1 1.3182 0.1775 0.9704 1.666 55.17 <.0001
interact 1 -0.7781 0.3064 -1.3787 -0.1775 6.45 0.0111
Scale 0 1 0 1 1
调用NLMIXED过程,拟合经典的logistic regression回归模型
proc nlmixed data=seeds;
/* Cassical logistic regression using NLMIXED */
title 'Classical logistic regression with NLMIXED';
parms b0=-0.55 b1=0.15 b2=1.32 b12=-0.78;
logitp=b0+b1*seed+b2*extract+b12*interact;
p=exp(logitp)/(1+exp(logitp));
model r ~ binomial(n,p) ;
run;
得到:
Parameter Estimates
Parameter Estimate Standard Error DF t Value Pr > |t| Alpha Lower Upper Gradient
b0 -0.5582 0.126 21 -4.43 0.0002 0.05 -0.8202 -0.2961 -0.00000229
b1 0.1459 0.2232 21 0.65 0.5203 0.05 -0.3182 0.61 -8.82E-07
b2 1.3182 0.1775 21 7.43 <.0001 0.05 0.9491 1.6872 -0.00000161
b12 -0.7781 0.3064 21 -2.54 0.0191 0.05 -1.4154 -0.1408 -6.61E-07
调用NLMIXED过程,拟合经典带随机截距的logistic regression回归模型
proc nlmixed data=seeds;
/* Logistic regression + RANDOM plate INTERCEPT */
title 'Logistic regression with random plate intercept (NLMIXED)';
parms b0=-0.55 b1=0.15 b2=1.32 b12=-0.78;
logitp=b0+b1*seed+b2*extract+b12*interact+alpha;
p=exp(logitp)/(1+exp(logitp));
random alpha ~ normal(0,varu) subject=plate out=seedmixed;
model r ~ binomial(n,p) ;
run;
得到:
Parameter Estimates
Parameter Estimate Standard Error DF t Value Pr > |t| Alpha Lower Upper Gradient
b0 -0.5484 0.1666 20 -3.29 0.0036 0.05 -0.896 -0.2009 -0.00087
b1 0.097 0.278 20 0.35 0.7308 0.05 -0.483 0.677 0.00022
b2 1.337 0.2369 20 5.64 <.0001 0.05 0.8428 1.8313 -0.00018
b12 -0.8104 0.3852 20 -2.1 0.0482 0.05 -1.6139 -0.007 0.00037
1.07 0.295 varu 0.05581 0.05 20 9 0.05 -0.0527 0.1643 0.001515
当然了winBUGS的强大之处并不在于此,而是在处理诸如GLME(有些文献称GLMM),空间数据模型等计算复杂的模型,之后还会继续讨论。

参考文献:
[1] Crowder M J (1978) Beta-binomial Anova for proportions. Applied Statistics. 27, 34-37.
[2] Breslow N E and Clayton D G (1993) Approximate inference in generalized linear mixed models. Journal of the American Statistical Association. 88, 9-25.
最后再送出一本书,供大家研究参考
Disease Mapping with WINBUGS and MLWin(djvu格式)
WinBUGS在统计分析中的应用 第二部分完

发表/查看评论